L’accumulo di prove suggerisce che le interruzioni nella segnalazione di fosforilazione/defosforilazione sono associate a comportamenti patologici delle cellule. I progetti Phospho-proteome hanno identificato quasi 300.000 residui fosforilati di Ser/Thr / Tyr nelle proteine cellulari . Parallelamente, il progetto genoma umano ha scoperto circa 500 protein chinasi. Tuttavia, un numero limitato di protein chinasi è stato assegnato a ciascun sito di fosforilazione.
Il test di fosforilazione in gel è stato un metodo conveniente per rilevare le attività della protein chinasi in campioni solubilizzati con SDS. Questo metodo è stato applicato per l’identificazione di CaMKII, MYPT1-K (o ZIPK) e altri . In questo protocollo di analisi, le protein chinasi vengono caricate su un gel SDS-poliacrilammide incorporato con un polipeptide di substrato e quindi sottoposte a rinaturazione, fosforilazione utilizzando ATP radioattivo, seguita da autoradiografia per rilevare il substrato fosforilato. Inoltre, l’autofosforilazione delle chinasi è stata rilevata sulla membrana di nitrocellulosa o fluoruro di polivinilidene (PVDF) utilizzando ATP radioattivo . Il materiale radioattivo può essere una limitazione per gli utenti, a causa delle norme di sicurezza. Inoltre, gli autoradiogrammi delle proteine fosforilate mancano di qualsiasi informazione sui siti di fosforilazione.
Qui, abbiamo ottimizzato un test alternativo nonradioattivo al tradizionale test di fosforilazione in gel. Denominato chinasi activity-tagged western blotting (KAT-WB), questo rileva con successo le Tyr-chinasi che si autofosforilano e le Ser/Thr-chinasi che fosforilano una proteina di substrato di interesse in un sito specifico utilizzando anticorpi specifici fosfo (P).
La figura 1 mostra i dati KAT-WB per il test di autofosforilazione Tyr-chinasi. Per realizzare macchie duplicate, due aliquote di un estratto sono state caricate su un gel di poliacrilammide SDS, sottoposto a elettroforesi, e quindi le proteine sono state trasferite su un foglio di membrana PVDF (0,2 µm). La membrana è stata trattata sequenzialmente con 2-propanolo tamponato, 6,0 M di guanidina cloridrato (Gu-HCl), 3,0 M di Gu-HCl, 0,1 M di Gu-HCl e tampone di rinaturazione (vedere Protocollo supplementare). Dopo una fase di rinaturazione notturna a 4°C, la membrana è stata tagliata longitudinalmente per generare due pezzi duplicati (Figura 1). Un pezzo di membrana è stato incubato per 60 min con 1 mm MgATP. L’altro pezzo di membrana è stato incubato senza MgATP, come controllo. Entrambi i fogli sono stati sottoposti contemporaneamente a immunodetezione con anticorpo anti-P-Tyr (Clone PY20 a diluizione 1:5000). Come mostrato nella Figura 1, sono state rilevate più bande P-Tyr a 100, 70, 45 e 35 kDa nella membrana di controllo (in alto a sinistra). Queste proteine P-Tyr erano presumibilmente già fosforilate prima dell’estrazione. L’incubazione con MgATP dopo la separazione ha elevato la fosforilazione di Tyr di una proteina a 45 kDa nei lisati del muscolo cardiaco (Figura 1, in alto a destra, corsia 2), indicando l’autofosforilazione dei residui di Tyr in questa chinasi sulla membrana dopo la rinaturazione. Figura 1 mostra densitogrammi di corsia 2 (- /+MgATP). Per verificare il carico equivalente di proteine sulle due macchie, i filtri sono stati sottoposti a colorazione Coomassie (Figura 1, in basso). Una nota tecnica è che, nel corso dello sviluppo del metodo, abbiamo scoperto che i trattamenti con 2-propanolo tamponato e 6M Gu-HCl erano entrambi necessari per la rilevazione delle attività chinasiche. Pertanto, KAT-WB ha rilevato una chinasi Tyr 45-kDa.

Le proteine totali del muscolo scheletrico murino (31 µg), del muscolo cardiaco (28 µg) e delle cellule Neuro2a (27 µg) sono state sottoposte a KAT-western blotting. Dopo la rinaturazione, la membrana è stata tagliata longitudinalmente in due e ciascuna parte è stata incubata per 60 min a 37°C in assenza (sinistra) o presenza (destra) di 1 mm ATP. La fosforilazione Tyr è stata rilevata con un metodo elettrochimiluminescente convenzionale utilizzando l’anticorpo anti-P-Tyr (top). Dopo l’immunoblotting, le proteine totali su ogni macchia sono state visualizzate dalla colorazione di Coomassie (in basso). I numeri a destra indicano i pesi molecolari delle proteine marker (in kDa). Una casella a destra indica densitogrammi della corsia 2 nei pannelli sinistro e destro. I livelli di luminosità e contrasto delle macchie sono identici.
Un’altra versione di KAT-WB utilizza la fosforilazione site-specific di una proteina di substrato assorbita sulla membrana PVDF in prossimità della chinasi. Abbiamo usato la fosfatasi holoenzyme inhibitor-1 (PHI-1) come proteina di interesse, la cui fosforilazione a Thr57 converte questa proteina cellulare in un inibitore della fosfatasi di tipo 1 Ser/Thr . La figura 2A mostra i dati di KAT-WB utilizzando come controllo positivo lo ZIPK ricombinante, una chinasi Ser / Thr nota per fosforilare il PHI-1 ricombinante a Thr57 . Dopo la denaturazione e il processo di rinaturazione graduale, la membrana è stata immersa durante la notte a 4 ° C nel tampone di rinaturazione integrato con PHI-1 wild type ricombinante (WT) o una versione non fosforilabile (T57H) a 0,2 mg/ml, e quindi la reazione di fosforilazione è stata eseguita con MgATP. Il PHI-1 fosforilato sulla membrana è stato rilevato usando l’anticorpo anti-P-PHI-1 (Thr57), e questo ha rivelato una banda dello ZIPK ricombinante. Tuttavia, solo un debole segnale è stato rilevato sulle membrane di controllo senza PHI-1 o con la versione T57H di PHI-1. Questi dati suggeriscono che le chinasi renature sulla membrana sono in grado di fosforilare i substrati che vengono anche assorbiti sulla membrana. KAT-WB è stato applicato per rilevare più chinasi PHI-1 nei tessuti e nelle cellule (Figura 2B). Estratti da cellule scheletriche, cardiache e Neuro2a di ratto sono stati sottoposti al test KAT-WB. Rispetto al controllo no-ATP (sinistra, in alto), i segnali di P-PHI-1 sono comparsi sulla macchia incubata con MgATP da 1 mm (destra, in alto). Bande prominenti di P-PHI-1 sono state rilevate nei lisati delle cellule di Neuro2a a 100, 60 e 49 kDa (corsia 3, punta di freccia aperta). Tuttavia, nessuna banda per P-PHI-1 era evidente nella corsia con gli estratti di muscoli scheletrici e cardiaci (corsie 1 e 2). Il segnale P-PHI-1 non è stato migliorato dalla fase di fosforilazione senza PHI-1 (Figura 2, in basso, vuoto: BLK). Questi risultati mostrano che ci sono più chinasi PHI-1 espresse nelle cellule neuronali ma non nei muscoli scheletrici o cardiaci. Questo ci fornisce un potente strumento per rilevare più chinasi specifiche del substrato in cellule e tessuti. Una nota tecnica è che la rinaturazione notturna con PHI-1 era necessaria per il rilevamento dei segnali di fosforilazione e che una maggiore concentrazione di PHI-1 non aumentava l’intensità della colorazione (non mostrata).
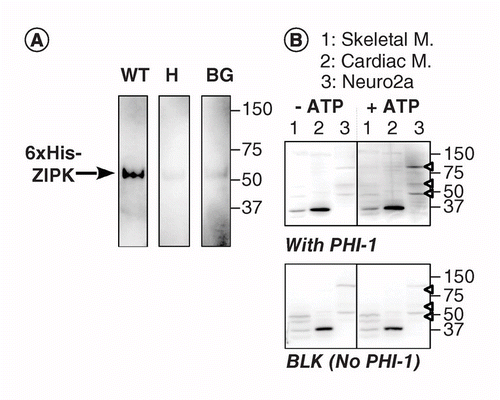
(A) Le macchie triplicate sono state preparate utilizzando ZIPK ricombinante parzialmente purificato da lisati di cellule che esprimono una versione con tag 6× His. Dopo l’elettroblotting e il processo di denaturazione-rinaturazione, la membrana è stata incubata durante la notte con proteine PHI-1 ricombinanti (WT: sinistra; T57H: centro) a 0,2 mg/ml o senza PHI-1 (BG). Ogni membrana è stata quindi incubata con ATP da 1 mm a 37°C e sottoposta a immunodetezione con anticorpo anti-P-PHI-1 (T57). B) Sono state preparate due macchie duplicate (superiore e inferiore) utilizzando campioni di proteine totali, come descritto nella figura 1. PHI-1 ricombinante (0,2 mg/ml) è stato aggiunto al tampone di rinaturazione per una macchia (in alto). BLK indica la macchia senza il trattamento ricombinante PHI-1. Dopo l’incubazione in assenza (sinistra) e presenza (destra) di ATP 1 mM, le macchie sono state sottoposte a immunodetezione utilizzando anti-P-PHI-1 (Thr57). Punte di freccia a destra indicano le posizioni di P-PHI-1 nel pannello in alto a destra. I livelli di luminosità e contrasto delle macchie sono identici.
BG: Sfondo; PESO: Wild-type.
Sulla base di questi dati, proponiamo che KAT-WB sia in grado di rilevare l’autofosforilazione delle Tyr chinasi e la fosforilazione sito-specifica delle proteine di substrato selettive di interesse da parte di più proteine chinasi presenti nei tessuti e nelle cellule. KAT-WB condivide essenzialmente i vantaggi del tradizionale test di fosforilazione in gel, che rileva le attività della chinasi in una miscela grezza, come gli estratti cellulari, esponendo le loro dimensioni molecolari . Un altro vantaggio di questi due saggi è che le modifiche post-traduzionali (PTMs) delle chinasi nelle cellule possono essere conservate in un momento desiderato in condizioni specifiche, perché i campioni per questi saggi vengono rapidamente denaturati con il tampone, inclusa la SDS, eliminando la fosforilazione e la defosforilazione negli estratti . La maggior parte delle proteine chinasi sono regolate attraverso PTM. KAT-WB è in grado di monitorare le fluttuazioni nelle attività della chinasi, anche se i PTM normativi devono ancora essere caratterizzati. KAT-WB non è in grado di rilevare ogni chinasi negli estratti cellulari perché solo gli enzimi monomerici che renature efficacemente nelle condizioni impiegate mostrano attività. Tuttavia, le modifiche di KAT-WB dovrebbero essere in grado di rilevare le chinasi regolate dalla calmodulina e dalle cicline, come è stato fatto mediante test di fosforilazione in gel . Il miglioramento dell’efficienza di rinaturazione e una combinazione con immunoprecipitazione/immunodepletion con anticorpi per chinasi possono espandere l’utilità dell’approccio.
È interessante notare che, oltre alla fosforilazione, il principio del protocollo KAT-WB può essere applicato per il rilevamento di altri enzimi PTM, come acetilasi, metilasi e ubiquitina ligasi, quando sono disponibili anticorpi specifici per PTM. Insieme con lo sviluppo di anticorpi che riconoscono PTMs site-specific, obiettivi di ricerca coperti da attività-tagged western blotting saranno ampliati.
Contributi dell’autore
M Eto, S Katsuki, Y Tanaka e K Takeya hanno concepito e progettato gli esperimenti. S Katsuki, Y Tanaka e K Takeya hanno condotto le analisi. M Eto, S Katsuki, Y Tanaka e K Takeya hanno eseguito l’analisi dei dati. M Eto, S Katsuki, Y Tanaka e K Takeya hanno scritto il giornale.
Ringraziamenti
Gli autori ringraziano DL Brautigan per la correzione del manoscritto e per i preziosi suggerimenti.
Finanziario& disclosure di interessi concorrenti
Questo lavoro è supportato da JSP KAKENHI (JP19K06573 e JP19H03125) e Società per la ricerca sul gusto Umami, fondi della città di Imabari e della prefettura di Ehime e un regalo di H Awagi. Gli autori non hanno altre affiliazioni rilevanti o coinvolgimento finanziario con qualsiasi organizzazione o entità con un interesse finanziario o conflitto finanziario con l’oggetto o materiali discussi nel manoscritto oltre a quelli divulgati.
Nella produzione di questo manoscritto non è stata utilizzata alcuna assistenza scritta.
Open access
Questo lavoro è concesso in licenza sotto la licenza Attribution-NonCommercial-NoDerivatives 4.0 Unported. Per visualizzare una copia di questa licenza, visitare http://creativecommons.org/licenses/by-nc-nd/4.0/
- 1. I nostri servizi sono a vostra disposizione. PhosphoSitePlus, 2014: mutazioni, PTMS e ricalibrazioni. Acidi nucleici Res. 43, D512–D520 (2015).Crossref, Medline, CAS, Google Scholar
- 2. Celenza JL, Carlson M. Rinaturazione dell’attività della protein chinasi sulle macchie proteiche. Metodi Enzimolo. Academic Press, Cambridge Massachusetts, 34, 423-430 (1991).Crossref, Google Scholar
- 3. Hutchcroft JE, Anostario M, Harrison ML et al. Renaturation and assay of protein kinases after electrophoresis in sodium dodecyl sulfate–polyacrylamide gels. Methods Enzymol. Academic Press,Cambridge Massachusetts, 33, 417–423 (1991).Crossref, Google Scholar
- 4. Kameshita I, Fujisawa H. A sensitive method for detection of calmodulin-dependent protein kinase II activity in sodium dodecyl sulfate-polyacrylamide gel. Anal. Biochem. 183(1), 139–143 (1989).Crossref, Medline, CAS, Google Scholar
- 5. Shackelford DA, Zivin JA. Rinaturazione dell’attività protein-chinasi calcio/calmodulina-dipendente dopo trasferimento elettroforetico da gel di sodio dodecil solfato-poliacrilammide alle membrane. Anale. Biochimica. 211(1), 131–138 (1993).Crossref, Medline, CAS, Google Scholar
- 6. Macdonald JA, Borman MA, Muranyi A et al. Identificazione della chinasi associata alla miosina fosfatasi della muscolatura liscia endogena. Proc. Natl Acad. Sic. Stati Uniti 98(5), 2419-2424 (2001).Crossref, Medline, CAS, Google Scholar
- 7. Wooten MW. Dosaggio della chinasi in-gel come metodo per identificare i substrati della chinasi. Sic. STKE 2002 (153), pl15 (2002).Medline, Google Scholar
- 8. Celenza J, Carlson M. Un gene del lievito che è essenziale per il rilascio dalla repressione del glucosio codifica una protein chinasi. Scienza 233(4769), 1175-1180 (1986).Crossref, Medline, CAS, Google Scholar
- 9. Eto M, Karginov A, Brautigan DL. Un nuovo inibitore della fosfoproteina degli oloenzimi della fosfatasi di tipo 1 della proteina. Biochimica 38(51), 16952-16957 (1999).Crossref, Medline, CAS, Google Scholar
- 10. Macdonald JA, Eto M, Borman MA et al. Doppia fosforilazione Ser e Thr di CPI-17, un inibitore della miosina fosfatasi, da parte della chinasi associata al MYPT. FEBS Lett. 493(2–3), 91–94 (2001).Crossref, Medline, CAS, Google Scholar
- 11. Eto M, Leach C, Tountas NA et al. Inibitori della fosfoproteina della proteina fosfatasi-1. Metodi Enzimolo. 366, 243–260 (2003).Medline, CAS, Google Scholar
- 12. Rousell J, Haddad El B, Lindsay MA et al. Regolazione dell’espressione genica del recettore muscarinico m2 da parte del fattore di crescita derivato dalle piastrine: coinvolgimento delle protein chinasi extracellulari regolate dal segnale nel processo di down-regulation. Mol. Pharmacol. 52(6), 966–973 (1997).Crossref, Medline, CAS, Google Scholar