La acumulación de evidencia sugiere que las interrupciones en la señalización de fosforilación/desfosforilación están asociadas con comportamientos patológicos de las células. Los proyectos de fosfoproteoma han identificado cerca de 300.000 residuos Ser/Thr/Tyr fosforilados en proteínas celulares . Paralelamente, el proyecto genoma humano descubrió aproximadamente 500 proteínas quinasas. Sin embargo, se ha asignado un número limitado de proteínas quinasas a cada sitio de fosforilación.
El ensayo de fosforilación en gel ha sido un método conveniente para detectar actividades de proteína quinasa en muestras solubilizadas con SDS. Este método se aplicó para la identificación de CaMKII, MYPT1-K (o ZIPK) y otros . En este protocolo de ensayo, las proteínas quinasas se cargan en un gel de poliacrilamida SDS incrustado con un polipéptido de sustrato, y luego se someten a renaturalización, fosforilación utilizando ATP radiactivo, seguido de autorradiografía para detectar sustrato fosforilado. Además, se ha detectado autofosforilación de quinasas en la membrana de nitrocelulosa o fluoruro de polivinilideno (PVDF) utilizando ATP radiactivo . El material radiactivo puede ser una limitación para los usuarios, debido a las normas de seguridad. Además, los autorradiogramas de proteínas fosforiladas carecen de información sobre los sitios de fosforilación.
Aquí, optimizamos un ensayo alternativo no radiactivo al ensayo tradicional de fosforilación en gel. Llamado western blotting con etiqueta de actividad cinasa (KAT-WB), detecta con éxito Tir-quinasas que se autofosforilan a sí mismas, así como Ser/Thr-quinasas que fosforilan una proteína de sustrato de interés en un sitio específico utilizando anticuerpos específicos de fosfo (P).
La figura 1 muestra los datos de KAT-WB para el ensayo de autofosforilación de Tir-quinasa. Para hacer manchas duplicadas, se cargaron dos alícuotas de un extracto en un gel de poliacrilamida SDS, se sometieron a electroforesis, y luego se transfirieron proteínas a una lámina de membrana de PVDF (0,2 µm). La membrana se trató secuencialmente con 2-propanol tamponado, 6,0 M de clorhidrato de guanidina (Gu-HCl), 3,0 M de Gu-HCl, 0,1 M de Gu-HCl y tampón de renaturalización (ver Protocolo Suplementario). Después de un paso de renaturalización durante la noche a 4 ° C, la membrana se cortó longitudinalmente para generar dos piezas duplicadas (Figura 1). Se incubó una pieza de membrana durante 60 min con MgATP de 1 mm. La otra pieza de membrana se incubó sin MgATP, como control. Ambas láminas se sometieron simultáneamente a inmunodetección con anticuerpo anti-P-Tyr (Clon PY20 a dilución de 1:5000). Como se muestra en la Figura 1, se detectaron múltiples bandas de P-Tyr a 100, 70, 45 y 35 kDa en la membrana de control (parte superior izquierda). Estas proteínas P-Tyr probablemente ya estaban fosforiladas antes de la extracción. La incubación con MgATP después de la separación elevó la fosforilación de Tyr de una proteína a 45 kDa en lisados de músculo cardíaco (Figura 1, arriba a la derecha, carril 2), indicando autofosforilación de residuos de Tyr en esta quinasa en la membrana después de la renaturalización. La Figura 1 muestra densitogramas del carril 2 (- /+MgATP). Para verificar la carga equivalente de proteína en las dos manchas, los filtros se sometieron a tinción de Coomassie (Figura 1, abajo). Una nota técnica es que, durante el desarrollo del método, encontramos que los tratamientos con 2-propanol tamponado y 6M Gu-HCl eran necesarios para la detección de las actividades de la quinasa. Por lo tanto, KAT-WB detectó una cinasa Tyr de 45 kDa.

Las proteínas totales del músculo esquelético de ratón (31 µg), el músculo cardíaco (28 µg) y las células Neuro2a (27 µg) se sometieron a la transferencia KAT-western. Después de la renaturalización, la membrana se cortó longitudinalmente en dos, y cada parte se incubó durante 60 min a 37°C en ausencia (izquierda) o presencia (derecha) de 1 mm de ATP. La fosforilación Tyr se detectó mediante un método electroquimioluminiscente convencional utilizando anticuerpos anti-P-Tyr (top). Después de la inmunoblocción, se visualizaron las proteínas totales en cada blot mediante tinción de Coomassie (parte inferior). Los números a la derecha indican los pesos moleculares de las proteínas marcadores (en kDa). Una caja a la derecha indica densitogramas del carril 2 en los paneles izquierdo y derecho. Los niveles de brillo y contraste de las manchas son idénticos.
Otra versión de KAT-WB utiliza fosforilación específica de un sitio de una proteína de sustrato absorbida en la membrana de PVDF en proximidad de la quinasa. Utilizamos el inhibidor de holoenzimas de fosfatasa-1 (PHI-1) como proteína de interés, cuya fosforilación en Thr57 convierte esta proteína celular en un inhibidor de la fosfatasa Ser/Thr tipo 1 . La Figura 2A muestra los datos de KAT-WB utilizando como control positivo ZIPK recombinante, una quinasa Ser / Thr conocida por fosforilar la PHI-1 recombinante en Thr57 . Después de la desnaturalización y el proceso de renaturalización escalonada, la membrana se empapó durante la noche a 4°C en el tampón de renaturalización suplementado con PHI-1 recombinante de tipo salvaje (WT) o una versión no fosforilable (T57H) a 0,2 mg/ml, y luego se ejecutó la reacción de fosforilación con MgATP. PHI-1 fosforilado en la membrana se detectó utilizando anticuerpos anti-P-PHI-1 (Thr57), y esto reveló una banda del ZIPK recombinante. Sin embargo, solo se detectó una señal débil en las membranas de control sin PHI-1 o con la versión T57H de PHI-1. Estos datos sugieren que las cinasas renaturadas en la membrana son capaces de fosforilar sustratos que también se absorben en la membrana. Se aplicó KAT-WB para detectar múltiples quinasas PHI-1 en tejidos y células (Figura 2B). Los extractos de esqueletos de ratas, músculos cardíacos y células Neuro2a se sometieron al ensayo KAT-WB. En comparación con el control sin ATP (izquierda, arriba), las señales de P-PHI-1 aparecieron en el blot incubado con MgATP de 1 mM (derecha, arriba). Se detectaron bandas prominentes de P-PHI-1 en lisados de células Neuro2a a 100, 60 y 49 kDa (carril 3, punta de flecha abierta). Sin embargo, no se evidenciaron bandas para P-PHI-1 en el carril con los extractos de músculos esqueléticos y cardíacos (carriles 1 y 2). La señal de P-PHI-1 no fue mejorada por el paso de fosforilación sin PHI-1 (Figura 2, abajo, en blanco: BLK). Estos resultados muestran que hay múltiples quinasas PHI-1 expresadas en las células neuronales, pero no en los músculos esqueléticos o cardíacos. Esto nos proporciona una herramienta poderosa para detectar quinasas múltiples específicas de sustrato en células y tejidos. Una nota técnica es que la renaturalización nocturna con PHI-1 era necesaria para la detección de las señales de fosforilación, y que una mayor concentración de PHI-1 no aumentaba la intensidad de la tinción (no se muestra).
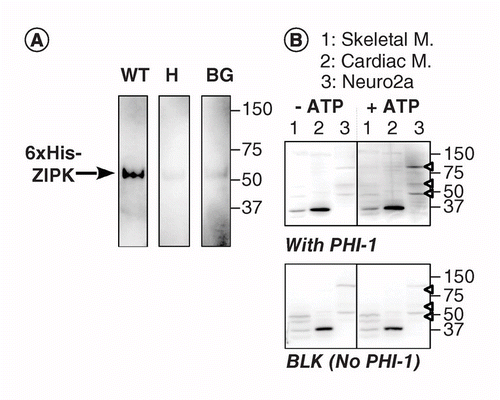
A) Se prepararon blots triplicados utilizando ZIPK recombinante parcialmente purificado a partir de lisados de células que expresaban una versión 6× marcada con His. Después del electroblot y del proceso de desnaturalización–renaturalización, la membrana se incubó durante la noche con proteínas recombinantes PHI-1 (peso: izquierda; T57H: centro) a 0,2 mg/ml o sin PHI-1 (BG). Cada membrana se incubó con ATP de 1 mM a 37 ° C y se sometió a inmunodetección utilizando anticuerpos anti-P-PHI-1 (T57). B) Se prepararon dos blots duplicados (superior e inferior) utilizando muestras totales de proteínas, como se describe en la Figura 1. Se añadió PHI-1 recombinante (0,2 mg/ml) al tampón de renaturalización para un blot (top). BLK indica el blot sin el tratamiento recombinante PHI-1. Después de la incubación en ausencia (izquierda) y presencia (derecha) de ATP de 1 mM, los blots se sometieron a inmunodetección utilizando anti-P-PHI-1 (Thr57). Las puntas de flecha a la derecha indican las ubicaciones de P-PHI-1 en el panel superior derecho. Los niveles de brillo y contraste de las manchas son idénticos.
BG: Fondo; WT: Tipo salvaje.
Con base en estos datos, proponemos que el KAT-WB es capaz de detectar la autofosforilación de cinasas Tir y la fosforilación de sitios específicos de proteínas de sustrato selectivo de interés por proteínas quinasas múltiples presentes en tejidos y células. KAT-WB comparte esencialmente las ventajas del ensayo tradicional de fosforilación en gel, que detecta las actividades de la quinasa en una mezcla cruda, como los extractos celulares, exponiendo sus tamaños moleculares . Otra ventaja de estos dos ensayos es que las modificaciones post-traslacionales (PTMs) de las quinasas en las células se pueden conservar en un punto de tiempo deseado bajo condiciones específicas, porque las muestras para estos ensayos se desnaturalizan rápidamente con el tampón, incluido el SDS, eliminando la fosforilación y desfosforilación en los extractos . La mayoría de las proteínas quinasas se regulan a través de la PTM. KAT-WB es capaz de monitorear las fluctuaciones en las actividades de la quinasa, incluso si los PMT reguladores aún no se han caracterizado. KAT-WB es incapaz de detectar cada quinasa en los extractos celulares porque solo las enzimas monoméricas que renaturalizan de manera efectiva bajo las condiciones empleadas muestran actividad. Sin embargo, las modificaciones de KAT-WB deben ser capaces de detectar quinasas reguladas por calmodulina y ciclinas, como se ha hecho mediante el ensayo de fosforilación en gel . La mejora de la eficiencia de renaturalización y una combinación con inmunoprecipitación/inmunodepresión con anticuerpos para quinasas pueden ampliar la utilidad del enfoque.
Es de destacar que, además de la fosforilación, el principio del protocolo KAT-WB se puede aplicar para la detección de otras enzimas PTM, como acetilasas, metilasas y ubiquitinas ligasas, cuando se dispone de anticuerpos específicos para PTM. Junto con el desarrollo de anticuerpos que reconocen los PTMS específicos del sitio, se ampliarán los objetivos de investigación cubiertos por el western blot con etiquetas de actividad.
Contribuciones de autor
M Eto, S Katsuki, Y Tanaka y K Takeya concibieron y diseñaron los experimentos. S Katsuki, Y Tanaka y K Takeya realizaron los ensayos. M Eto, S Katsuki, Y Tanaka y K Takeya realizaron el análisis de los datos. M Eto, S Katsuki, Y Tanaka y K Takeya escribieron el artículo.
Agradecimientos
Los autores agradecen a DL Brautigan por la revisión del manuscrito y por sus valiosas sugerencias.
Financiera & divulgación de intereses contrapuestos
Este trabajo cuenta con el apoyo de JSPS KAKENHI (JP19K06573 y JP19H03125) y la Sociedad para la Investigación del Sabor Umami, fondos de la ciudad de Imabari y la prefectura de Ehime, y un regalo de H Awagi. Los autores no tienen ninguna otra afiliación o participación financiera relevante con ninguna organización o entidad con un interés financiero o conflicto financiero con el tema o los materiales discutidos en el manuscrito, aparte de los divulgados.
No se utilizó ayuda de escritura en la producción de este manuscrito.
Acceso abierto
Este trabajo está licenciado bajo la Licencia Attribution-NonCommercial-NoDerivatives 4.0 Unported. Para ver una copia de esta licencia, visite http://creativecommons.org/licenses/by-nc-nd/4.0/
- 1. Hornbeck PV, Zhang B, Murray B et al. PhosphoSitePlus, 2014: mutaciones, PTMs y recalibraciones. Ácidos nucleicos Res. 43, D512–D520 (2015).Crossref, Medline, CAS, Google Scholar
- 2. Celenza JL, Carlson M. Renaturalización de la actividad de la proteína quinasa en las manchas de proteína. Métodos Enzymol. Academic Press, Cambridge, Massachusetts, 34, 423-430 (1991).Crossref, Google Scholar
- 3. Hutchcroft JE, Anostario M, Harrison ML et al. Renaturation and assay of protein kinases after electrophoresis in sodium dodecyl sulfate–polyacrylamide gels. Methods Enzymol. Academic Press,Cambridge Massachusetts, 33, 417–423 (1991).Crossref, Google Scholar
- 4. Kameshita I, Fujisawa H. A sensitive method for detection of calmodulin-dependent protein kinase II activity in sodium dodecyl sulfate-polyacrylamide gel. Anal. Biochem. 183(1), 139–143 (1989).Crossref, Medline, CAS, Google Scholar
- 5. Shackelford DA, Zivin JA. Renaturalización de la actividad de la proteína quinasa dependiente de calcio/calmodulina después de la transferencia electroforética de geles de sulfato de dodecilo de sodio y poliacrilamida a las membranas. Anal. Bioquímica. 211(1), 131–138 (1993).Crossref, Medline, CAS, Google Scholar
- 6. Macdonald JA, Borman MA, Muranyi A et al. Identificación de la quinasa asociada a la miosina fosfatasa del músculo liso endógeno. Proc. Natl Acad. Sci. USA 98(5), 2419-2424 (2001).Crossref, Medline, CAS, Google Scholar
- 7. Wooten MW. Ensayo de cinasa en gel como método para identificar sustratos de cinasa. Sci. STKE 2002 (153), pl15 (2002).Medline, Google Scholar
- 8. Celenza J, Carlson M. Un gen de levadura que es esencial para la liberación de la represión de la glucosa codifica una proteína quinasa. Ciencia 233(4769), 1175-1180 (1986).Crossref, Medline, CAS, Google Scholar
- 9. Eto M, Karginov A, Brautigan DL. Un nuevo inhibidor de fosfoproteínas de las holoenzimas de fosfatasa de proteína tipo 1. Bioquímica 38(51), 16952-16957 (1999).Crossref, Medline, CAS, Google Scholar
- 10. Macdonald JA, Eto M, Borman MA et al. Fosforilación dual de Ser y Thr de CPI-17, un inhibidor de la fosfatasa de miosina, por quinasa asociada a MYPT. FEBS Lett. 493(2–3), 91–94 (2001).Crossref, Medline, CAS, Google Scholar
- 11. Eto M, Leach C, Tountas NA et al. Inhibidores de fosfoproteínas de la proteína fosfatasa-1. Métodos Enzymol. 366, 243–260 (2003).Medline, CAS, Google Scholar
- 12. Rousell J, Haddad El B, Lindsay MA et al. Regulación de la expresión génica del receptor muscarínico m2 por factor de crecimiento derivado de plaquetas: participación de proteínas quinasas reguladas por señales extracelulares en el proceso de regulación descendente. Mol. Pharmacol. 52(6), 966–973 (1997).Crossref, Medline, CAS, Google Scholar